Populations of In Silico Models for the simulation of experimental variability
A seminar by Michelangelo Paci from Tampere University
-
Date: 15 DECEMBER 2017 from 9:00 to 12:00
-
Event location: DEI, Room A2, via Rasi e Spinelli, Cesena
-
Type: Seminar
Curriculum: Bioengineering
Registration
Registration is not compulsory, but highly recommended.
It will be possible to register until december 12 at 3 pm
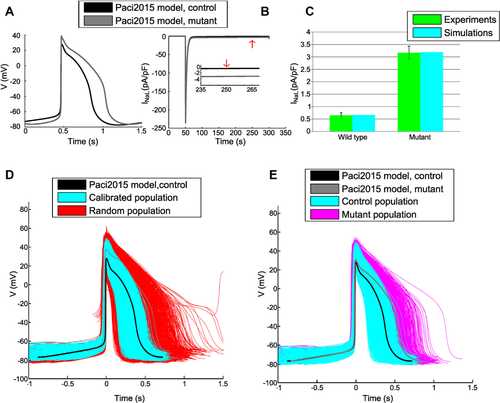
In this seminar, the basics for the design and implementation of populations of computational models of the cardiac action potential will be presented. In spite of the established usefulness of models of action potential for in silico studies of cardiac electrophysiology and for the assessment of the effects of drugs, a single computational model fails, due to its intrinsic limitations, to simulate the experimental variability which is always observed in vitro on cardiomyocytes.
A recent approach to overcome this limitation is to develop a population of models appropriately calibrated with experimental data, i.e. a set of hundreds/thousands of models appropriately selected to be in agreement with the experimental data. During the seminar an approach will be presented to generate populations of computational models, in particular i) how to sample the parameter space to generate a random population of models and ii) how to filter this population with measures of biomarkers calculated on the experimentally recorded action potentials. The specific kind of cells that will be considered to build the population of in silico models are cardiomyocytes derived from induced pluripotent human stem cells (hiPSC-CM): this particular cell type is in fact known for its extreme electrophysiological variability, which is reflected in a variety of different morphologies of action potential experimentally recorded in the lab.
----
In questo seminario, verranno presentate le basi necessarie alla progettazione e realizzazione di popolazioni di modelli computazionali del potenziale d’azione cardiaco. A dispetto della ormai comprovata utilità di modelli del potenziale d’azione per studi in silico dell’elettrofisiologia cardiaca e valutazione degli effetti di medicinali su di essa, un singolo modello computazionale non riesce, per i sui stessi limiti intrinseci, a simulare la variabilità sperimentale che viene osservata quotidianamente in vitro sui cardiomiociti.
Un recente approccio per superare questo limite consiste nello sviluppare una popolazione di modelli opportunamente calibrata con dati sperimentali, ossia un insieme di centinaia di modelli opportunamente selezionati per essere in accordo con i dati sperimentali. Nel corso del seminario verrà presentata un approccio per generare popolazioni di modelli computazionali, in particolare i) come campionare lo spazio dei parametri per generare una popolazione random di modelli e ii) come filtrate tale popolazione con misure di biomarker calcolate sui potenziali d’azione misurati su una popolazione in vitro di cardiomiociti. Le cellule considerate per costruire la popolazione di modelli in silico saranno i cardiomiociti derivati da cellule staminali umane pluripotenti indotte (hiPSC-CM): questo particolare tipo cellulare è infatti noto per l’estrema variabilità elettrofisiologica, che si riflette in una varietà di forme diverse di potenziale d’azione sperimentalemente registrate in laboratorio.
Contacts
Stefano Severi
Highlights
- Registration form Published